Thème de recherche n°2 : Régulation de la stabilité des ARNm par les ARN régulateurs chez Bacillus subtilis
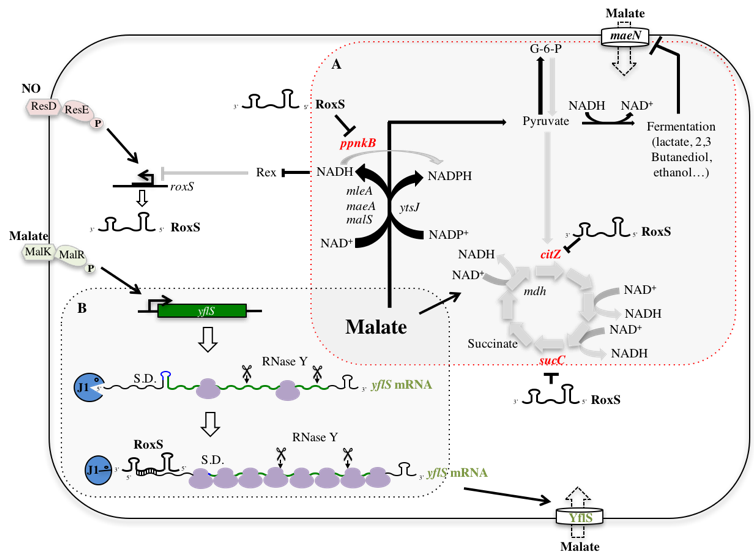
De nombreuses études transcriptionnelles ont permis de montrer que les bactéries à Gram-positifs possède une centaine d'ARN régulateur. Cependant, très peu de ces ARN régulateurs ont été caractérisés chez B. subtilis. Comme pour la dégradation des ARN, les ARNs non-codants ont principalement été étudiés chez les bactéries à Gram-négatif comme E. coli. La plupart des ARN étudiés jusqu'à récemment bloque la traduction des ARNm en s'hybridant avec la région de fixation du ribosome. Cependant, des études récentes montrent que les ARN régulateurs peuvent modifier la stabilité de certains ARN sans affecter leur traduction, en recrutant ou en interférant directement avec la machinerie de dégradation des ARN de la bactérie. Chez l'organisme modèle des bactéries à Gram-positif, B. subtilis, seulement quelques ARN non-codant ont été caractérisés et leur mécanisme de régulation reste largement méconnus. A partir des quelques études réalisées précédemment, il est clair que les mécanismes de régulation par les ARN non-codants chez les firmicutes sont différents de ceux trouvés chez E. coli. En effet, la protéines Hfq, qui joue un rôle primordial chez E. coli pour favoriser les interactions entre ARN non-dans et ARNm cibles, n'est pas requise chez B. subtilis. De plus, la RNase E, responsable de la dégradation des ARNm cibles après appariement avec leur ARN régulateurs chez E. coli, n'existe pas chez B. subtilis. Dans le but de mieux comprendre le mode de fonctionnement des ARN régulateurs chez B. subtilis, nous nous intéressons à leur caractérisation et à leurs interactions avec la machinerie de dégradation. C'est dans ce contexte que nous avons récemment caractérisé l'ARN régulateur RoxS, conservé chez la bactérie pathogène S. aureus (et appelé RsaE). Nous avons montré que la transcription de cet ARN est directement régulé par 2 facteurs de transcription: 1. Le système à 2 composantes ResD-ResE, qui répond a la présence de nitric oxide (NO) ou d'oxygène dans le milieu. Le facteur de transcription ResD active la transcription de RoxS. 2. l'inhibiteur de transcription Rex qui réagit au ratio NAD:NADH de la cellule. Nous avons aussi montré que RoxS impact l'expression de plus de 35 ARNm dans la cellule dont la majorité sont lié à la réponse au stress oxidant. Plusieurs gènes du cycle de Krebs sont aussi régulé par RoxS, notamment la succinate déhydrogénase (SucC). Dans ce cas, RoxS s'apparie à la région de fixation du ribosome pour bloquer l'initiation de la traduction de cet ARNm. Nous avons aussi montré que RoxS était capable d'interférer directement avec la dégradation de l'ARNm yflS, codant un transporteur du malate. En effet, l'appariement de RoxS à l'extrémité 5' non-traduite de cet ARN empêche l'exoribonucléase J1 d'attaquer cet ARNm.